用户名:
密码:

使用Infinite200酶标仪测定核酸浓度的方法
一、光吸收法
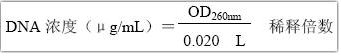
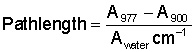
DNA的紫外吸收高峰为260nm,吸收低峰为230nm,而蛋白质的紫外吸收高峰为280nm。
如OD260nm/ OD230nm≥2,OD260nm/ OD280nm≥1.8,表示RNA 已经除净,蛋白含量不超过0.3%。
用这种方法适合测定GC含量比较均匀的基因组DNA,人工合成的寡核苷酸如果CG含量过大或者过小,就不能采用此方法。
双链DNA浓度的计算:
OD260nm为260nm 处的光吸收值;L 为光程(cm);0.020 为1μg/mL DNA 钠盐的光密度。
由于这种方法无法区分核酸种类和核苷酸,故此对于不纯的样品准确性不高,如果需要精确定量双链DNA的浓度,请采用荧光染料测定DNA的方法。
(一) 使用酶标板:
优点:通量大;
缺点:不易获得准确的光程;需要较高的样品浓度和较大的样品体积。
使用酶标板检测DNA,必须使用能够透过紫外光的UV板,普通酶标板是不能使用的。
市场上常见的两个品牌Costar和Greiner都有UV板,它们是有区别的:Costar的UV板只能做260nm-280nm的光吸收检测,波长低于260nm时板的本底光吸收会随波长降低不断增加;Greiner的UV Star板在230nm-280nm范围内吸收值大致相同,故此可应用范围更宽泛。
UV板在260nm有大约0.05OD的光吸收,按100ul的检测体积计算,相当于10ug /ml dsDNA,按照酶标仪检测的一般原则,样品的OD值需要为本底的3倍以上才可信,就是核酸样品的260nm光吸收值必须大于0.150OD,这样就要求100ul核酸样品的浓度不低于30ug/ml,如果检测体积减小,这个浓度还需要继续加大。
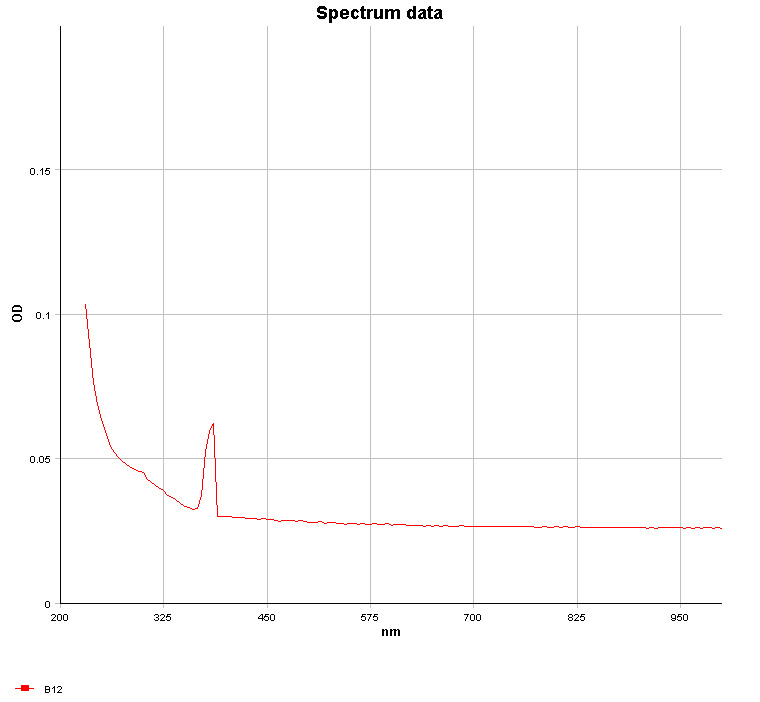
图1:Costar96孔UV板的光吸收谱
1、酶标板的选择:
DNA样品的体积一般是有限的,如果过度稀释将无法在酶标仪上检测。要避免过度稀释,可使用384孔UV板进行检测,需要15ul-20ul样品直接检测或稀释数倍后检测,也可使用half area 96孔UV板进行检测,需要20ul-80ul的样品,样品浓度仍需大于30ug/ml。
Costar和Greiner都提供384孔和half area 96孔UV板:
384:Greiner UV-Star Cat.No. 781801;Costar UV Cat.No. 3675;
Half area 96:Greiner UV-Star Cat.No. 675801;Costar UV Cat.No. 3679。
其中我们推荐使用Greiner UV-Star 96孔Half area板,因为它是一种可确定光程的板,80ul样品的光程是0.5cm,170ul样品的光程是1cm。此种板国内一般无库存,需要提前订货。

图2:Greiner UV-Star 96Well Half area 的几何尺寸
2、光程的确定
光程就是测定时光线穿过样品的距离,它与测量获得的光吸收值(OD值)成正比,要计算核酸浓度,必须知道测量的光程。由于酶标板的孔一般是半圆锥型的,上下截面的直径是不等的,所以使用酶标板测量DNA时,样品的光程是难以计算的。
常用的酶标板光程计算,是利用水在977nm或998nm下的吸收峰,来计算水溶液的光程。水在977nm-998nm有吸收峰,在900nm几乎无吸收。先利用分光光度计测出1cm光程的水在977nm或998nm和900nm的光密度差,再与酶标板上测得的差值比较,就可以估算出每个孔的光程了。
下面是光程计算公式:
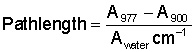
A977:水溶液样品在977nm的吸光值
A900:水溶液样品本底
Awatercm-1:1cm光程比色杯中水的吸光度值( A977 )-本底值( A900 )
光程测量法只能在光栅型酶标仪上使用,比如Infinite M200/M1000,由于水在977nm或998nm 下的吸收值本来就不大,这种方法误差相对来说就比较大。
也可以采用标准曲线法计算样品浓度从而避免测量光程;或者采用某个已知浓度、特征吸收峰和光密度的样品去估算光程(可以和分光光度计的测值比对,或者通过核酸浓度进行换算)。
(二) 使用比色皿:
优点:对样品浓度的要求低;
缺点:需要较大的样品体积,通量低。
如果M200酶标仪具有比色杯模块,可使用微量石英比色皿测定样品核酸浓度。由于M200的比色杯模块测量狭缝的尺寸限制,样品体积一般不能低于100ul,光学性能好的石英比色皿260nm本底吸收在0.01-0.04OD,大约相当于0.5-2.0 ug /ml dsDNA,低浓度的DNA样品可测量。
如果酶标仪不具备比色杯模块,也可以使用卧式微量石英比色皿来检测DNA,Tecan的酶标仪配置了卧式比色皿的适配器,但卧式比色皿需要的样品体积较大,需要200-400ul。
(三) Nanoquant板
优点:需要样品少,通量较大;
缺点:样品浓度不可过低。
NanoQuant是Tecan公司开发的利用Infinite200酶标仪光吸收测量功能检测微量体积(2ul)核酸样品的工具模块,样品不需要稀释直接检测,每次最多可检测16个样品。
NanoQuant板在260nm的本底吸收约为0.05OD,NanoQuant模块的检测下限为1ug /ml dsDNA。
NanoQuant模块包括NanoQuant板一块、辅助加样器一个。

图3:NanoQuant模块
Nanoquant模块的使用方法另作详述。
二、荧光法
使用与核酸结合的荧光染料测定DNA。荧光方法灵敏度高,特异性好,如PicoGreen灵敏度可达250pg/ml,能区分DNA和RNA,区分核酸和核苷酸,需要样品量少。荧光法的缺点是需要做标准曲线,荧光染料需要一定的成本,得不到A260nm/ A280nm值。
常用染料品种:
PicoGreen:Invitrogen生产的dsDNA定量染料Kit:(Molecular Probes P-7589)。
SYBRGreen I:激发波长497nm,发射波长520nm,用于核酸凝胶电泳染色和荧光定量PCR,价格便宜,直接用于酶标检测DNA本底较PicoGreen高,灵敏度需实验确定。
附:
DNA的SYBR Green I荧光定量方法
原理: SYBR Green I荧光染料与 DNA双链具有高亲和力,结合后荧光强度可增加800-1000倍;在溶液中SYBR Green I过量时,荧光强度与双链DNA浓度成正比。因此可用于溶液中 dsDNA 的定量,为核酸的高灵敏度检测提供了一种新的方式。
方法:
1. 将SYBR Green I储存液(10000×,Molecular Probes,catalog number S7563)用DMSO稀释至200×,再用1×TE(10mM Tris-Cl,1mM EDTA,pH 7.5)稀释至2×;
2. DNA标准品的稀释:已知浓度的纯化的DNA均可作为标准品。将标准DNA稀释为三个浓度:10ng/ml,100ng/ml,1000ng/ml。若能根据经验估计出待测样品DNA的大致浓度,则省去4-6步。
3. 待测DNA的稀释:将待测样品用1×TE进行梯度稀释,稀释2-3个浓度。
4. 加样:黑色平底96孔板(GREINER,)的每孔中加入100μl 2×SYBR Green I和100μl标准品稀释液(或待测DNA稀释液,以TE代替样品作为空白对照),混匀;室温避光孵育2-5分钟。
5. 放入TECAN多功能酶标仪中,设置好参数进行测量,测量结果保存,进行数据分析。
6. 根据减去背景后的荧光值估计出样品DNA的大致浓度(荧光值与浓度成正比),将标准DNA在适当范围内进行梯度稀释。
7. 重复4-6步测量。
注意事项:
1. 标准DNA和样品的稀释度均应在1×SYBR Green I测量的线性范围内(10-1000ng/ml),否则测量结果不准确;样品浓度在1-10ng/ml范围内时,用终浓度为0.2×的SYBR Green I测量;DNA浓度<1ng/ml时,不宜用SYBR Green I方法测量。
2. SYBR Green I的稀释应在塑料容器中进行,不要在玻璃容器中进行,因为玻璃表面可吸附染料;SYBR Green I见光易分解,因此工作液要避光,而且在稀释后的几个小时内尽快用完,不要长期保存。
3. 用来作标准曲线的DNA与待测DNA的纯化方式应尽量相同,以确保溶液内其他组分(盐、有机溶剂、蛋白等)一致,使测量准确。
4. 较高的样品DNA稀释浓度可减小其他污染(盐、有机溶剂、蛋白等)的干扰。
准备的试剂:SYBR Green I(200×)100 μl
1. 根据用量将SYBR Green I用蒸馏水稀释至1×。
2. 加样:每孔加入90μl 1×SYBR Green I和10μl待测样品溶液或标准品溶液,先根据标准品浓度判断样品的大体浓度范围。若样品浓度很低(1-10ng/ml)则用0.2×SYBR Green I测量。
